储俊东 刁澎云 李旭光 毕可然 周刚 周军 邓燕飞 许郑超


摘要:以中华绒螯蟹(Eriocheir sinensis)为试验对象,克隆中华绒螯蟹表皮蛋白CAP基因序列,分析其表达规律,为探索中华绒螯蟹蜕皮发生机理提供参考。运用生物信息学从中华绒螯蟹转录组数据中比对筛查表皮蛋白CAP基因序列信息,克隆扩增获得表皮蛋白CAP基因cDNA序列,运用RT-qPCR分析表皮蛋白CAP基因在不同组织、不同发育阶段和不同蜕皮时期的表达特征。结果表明,中华绒螯蟹表皮蛋白CAP基因cDNA序列全长380 bp,编码101个氨基酸,包括1个信号肽和1个R&R结合域。表皮蛋白基因CAP主要在蜕皮后期的表皮组织中表达,在蜕皮间期不表达;在不同发育阶段的仔蟹1期表达量最高,根据其发育表达模式推测表皮蛋白基因CAP参与表皮的形成和钙化,为后续深入研究表皮蛋白基因的生理功能奠定基础。
关键词:中华绒螯蟹;表皮蛋白;基因克隆;mRNA表达
中图分类号:
S917文献标志码:
A
文章编号:1002-1302(2021)08-0074-06
收稿日期:20200-08-11
基金项目:国家现代农业产业技术体系建设专项(编号:CARS-48);江苏省自然科学基金面上项目(编号:BK20161602);江苏省渔业科技类重点项目(编号:D2018-4);江苏省农业重大新品种创制项目(编号:PZCZ201748);江苏省现代农业(中华绒螯蟹)产业技术体系项目(编号:JATS[2019]386);江苏省研究生科研与实践创新计划(编号:SJCX20_1269、SJCX20_1293)。
作者简介:储俊东(1996—),男,江苏南通人,硕士研究生,主要从事水生甲壳类分子遗传学研究。E-mail:qichenchu1@gmail.com。
通信作者:李旭光,副研究员,主要从事水生甲壳类繁育与生态养殖研究。Tel:(025)86851575;E-mail:xuguangli1981@163.com。
甲壳动物的体表包裹着1层厚厚的外骨骼,它不但保护着甲壳动物免受外来细菌和病毒的侵袭,还在其维持正常生命活动和体态的建成中均发挥着非常重要的作用。甲壳类表皮由外向内依次可分为上表皮、外表皮、内表皮及真皮细胞,其中内表皮是表皮皮层中的主要组成部分,主要由几丁质、表皮蛋白及钙盐等构成[1]。坚硬外骨骼虽然极大地增强了甲壳动物的生存和适应能力,但也限制了其生长。因此,甲壳类需要周期性地蜕去旧表皮,合成新表皮。甲壳类动物的蜕皮周期主要分为蜕皮前期(D)、蜕皮期(E)、蜕皮后期(A、B)和蜕皮间期(C)4个阶段。蜕皮前期(D),在蜕皮类调控激素的诱导下旧表皮被几丁质代谢酶类及蛋白水解酶类部分消化和再吸收,新表皮开始逐渐分泌形成;蜕皮期(E),机体旧表皮褪去,新表皮替代舊表皮;蜕皮后期(A、B),机体吸收大量水分迅速增长增质量,同时表皮组织迅速钙化;当表皮钙化终止,机体进入蜕皮间期(C)[2-3]。
表皮蛋白是构成甲壳类表皮的重要结构蛋白,不同类型的表皮蛋白与长链几丁质相结合,从而影响表皮结构及其性能。早期由于传统的提取分离纯化方法的局限性限制了对表皮蛋白的发掘,因此有关表皮蛋白种类与数量的报道较少。近年来,随着基因组、转录组等测序技术的兴起,大大加速了人们对于表皮蛋白的发现,目前已在一些模式昆虫如黑腹果蝇(Drosophila melanogaster)、冈比亚按蚊(Anopheles gambiae)、家蚕(Bombyx mori)及一些重要经济甲壳类如南美白对虾(Penaeus vannamei)、红螯螯虾(Cherax quadricarinatus)中鉴别出大量表皮蛋白基因家族[4-5]。其中,具有与几丁质相结合R&R结构域(rebers & riddiford consensus)的CPR家族数量多、分布广,是迄今为止表皮蛋白家族中含量最丰富的一个家族。根据R&R结构域的保守型序列,CPR家族又可细分为RR-1、RR-2和 RR-3 等3 个亚家族。其中,RR-1亚族主要存分布于柔软的未钙化表皮;而RR-2亚族主要存在于坚硬的钙化表皮;RR-3家族数量最少,其分布特征还不太明确[6-7]。值得注意的是,在克氏原螯虾(Procambarus clarkii)中发现的一种特殊的钙相关蛋白CAP-1和CAP-2,序列分析发现其属于CPR家族,同时具有几丁质结合能力和抑制碳酸钙沉淀的能力,这种具有多重功能的表皮蛋白是现在研究的重点。
中华绒螯蟹(Eriocheir sinensis)又称河蟹,是重要的经济养殖蟹类,广泛分布于我国东南沿海以及长江、瓯江、辽河水域。江苏是长江流域中华绒螯蟹的发源地和故乡,跨江临海,水网密布,河湖众多,有着得天独厚的发展河蟹繁育与养殖的自然条件。2018年,江苏河蟹养殖面积2 666.67 km2,年产量32 万t,总产值270 亿元,总产量和总产值均占全国总量的1/2,河蟹养殖业成为江苏渔业的优势产业[8]。目前,有关甲壳类表皮蛋白的研究主要集中在虾类和鳌虾类上,有关短尾派中华绒螯蟹R&R型表皮蛋白还鲜有报道。本试验通过转录组结合分子克隆获得中华绒螯蟹表皮蛋白CAP(calcification-associated peptide)cDNA序列,命名为EsCAP,采用定量PCR检测EsCAP在不同组织的分布,分析EsCAP在不同发育阶段以及蜕皮周期的表达规律,为后续进一步研究其在蜕皮生长中的功能作用奠定了基础。
1 材料与方法
1.1 研究对象
2019年4月从江苏省淡水水产研究所扬中基地挑取健康中华绒螯蟹1龄幼蟹作为试验样本,运至江苏省淡水水产研究所实验室室温下暂养并开展后续试验,水深50 cm,使用空气泵不间断通氧,确保养殖时箱内水体溶氧。试验开展期间采用人工饲料饲喂,每晚喂食,早上对水族箱底部污物进行及时的清理,保证养殖水环境清洁。
1.2 主要试剂
Total RNA提取试剂盒和反转录试剂盒购自TaKaRa公司,PCR试剂购自生工生物(上海)股份有限公司,qPCR试剂(SYBR Premix Ex TaqTM Ⅱ 和荧光染料 ROX) 购自宝生物工程(大连)有限公司,DNA marker、PUCm-T载体、琼脂糖凝胶回收试剂盒等购自南京诺唯赞生物科技股份有限公司,其他试剂为国产分析纯。
1.3 组织样品的收集
参考甲壳类分期方法,根据中华绒螯蟹壳色与硬度等形态学特征分为蜕皮前期(D)、蜕皮后期(A、B期)、蜕皮间期(C期)4个蜕皮时期。蜕皮前期(D),外表皮内陷,新的外表皮建逐渐形成,新生刚毛逐步从旧刚毛中分离;蜕皮后期(A期),中华绒螯蟹刚刚从老旧的壳中挣脱出来,新生壳柔软黑亮而有弹性;蜕皮后期(B期),新生表皮开始钙化,新壳逐渐硬化,体长固定;蜕皮间期(C期),新表皮钙化完成,中华绒螯蟹开始大量摄食,进行营养积累,为下次蜕皮做准备。分别采集中华绒螯蟹蜕皮间期与蜕皮后期(B期)表皮、肝胰腺、肌肉、鳃、眼柄、附肢、心脏、肠道、胃、血淋巴共10个组织样本。在解剖样品前先对样品进行清洗并用冰袋降低中华绒螯蟹的活跃度。采集中华绒螯蟹受精卵、溞1期(ZⅠ)、溞3期(ZⅢ)、溞5期(ZⅤ)、大眼幼体和仔蟹1期6个不同发育阶段的个体样本。所有收集样本经液氮冷却后放在-80 ℃冰箱保存备用。
1.4 总RNA的提取与cDNA的逆转录
用TaKaRa Mini BEST Universal RNA Extraction Kit试剂盒提取中华绒螯蟹各组织、不同蜕皮时期表皮和不同发育阶段个体总RNA。提取出的总RNA用1%琼脂糖凝胶电泳检测其质量,并用紫外分光光度计检测其浓度,测完后立即置于-80 ℃保存备用。逆转录反应按照PrimeScriptTM RT reagent Kit with gDNA Eraser (Perfect Real Time)试剂说明书进行,获得第1链cDNA,用于RT-qPCR反应,合成之后置于-20 ℃保存备用。
1.5 设计引物
利用在线设计引物软件Primer 3 input设计引物,扩增全长引物EsCAP F1/R1,由表1可知PCR反应条件为:94 ℃ 30 s;55 ℃ 30 s,72 ℃ 1 min,30 个循环;72 ℃延伸10 min。配制1%琼脂糖凝胶对PCR产物进行检测,回收所需DNA片段,之后按照FastPureGel DNA Extraction Mini Kit试剂盒进行纯化操作。纯化后的产物与PUCm-T载体结合,将结合产物导入感受态细胞中进行蓝白斑筛选,挑取目的菌落。经PCR筛选后送到生工生物(上海)股份有限公司测序。获得EsCAP开放阅读框(ORF)后,再设计定量引物RT-EsCAP F2/R2并用以进行RT-qPCR。
1.6 RT-qPCR检测
以β-actin基因为内参基因,无菌超纯水为阴性对照,采用RT-qPCR(SYBR Green)检测表皮蛋白CAP在不同组织、不同蜕皮时期和不同发育阶段的表达量。RT-qPCR反应程序:95 ℃预变性 5 min,1次循环;PCR反应95 ℃ 30 s,60 ℃ 20 s,40 个循环。反应结束后获得扩增曲线。反应在LightCycler480荧光定量PCR仪上进行,设cDNA样品3次重复,反应结束后采集目标基因的CT值(C表示循环数;T表示荧光阈值)和内参基因的CT值,qRT-PCR结果采用2-ΔΔCT法分析,数据处理和作图使用Excel和Sigma Polt 14.0软件,结果均以“x±s”表示。采用SPSS软件的邓肯法分析EsCAP表达量是否具有显著差异。
1.7 生物信息学分析
用DNA MAN 软件将所得的DNA序列翻译成蛋白序列,用Edit seq软件对蛋白序列理化性质和相对分子质量进行预测,用NCBI在线工具ORF finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)搜索EsCAP开放阅读框;从NCBI数据库中用BLASTP程序检索氨基酸同源性序列,并用Clustalw2和Genedoc软件对中華绒螯蟹表皮蛋白EsCAP基因和检索出的氨基酸序列进行同源性比对,用MEGA 6.0软件构建进化树,用DNA MAN对其进行序列一致性分析,使用在线软件SMART(http://smart.embl-heidelberg.de/)进行氨基酸序列的结构域分析,使用cuticleDB(http://bioinformatics2.biol.uoa.gr/cuticleDB/index.jsp)在线分析R&R序列。
2 结果与分析
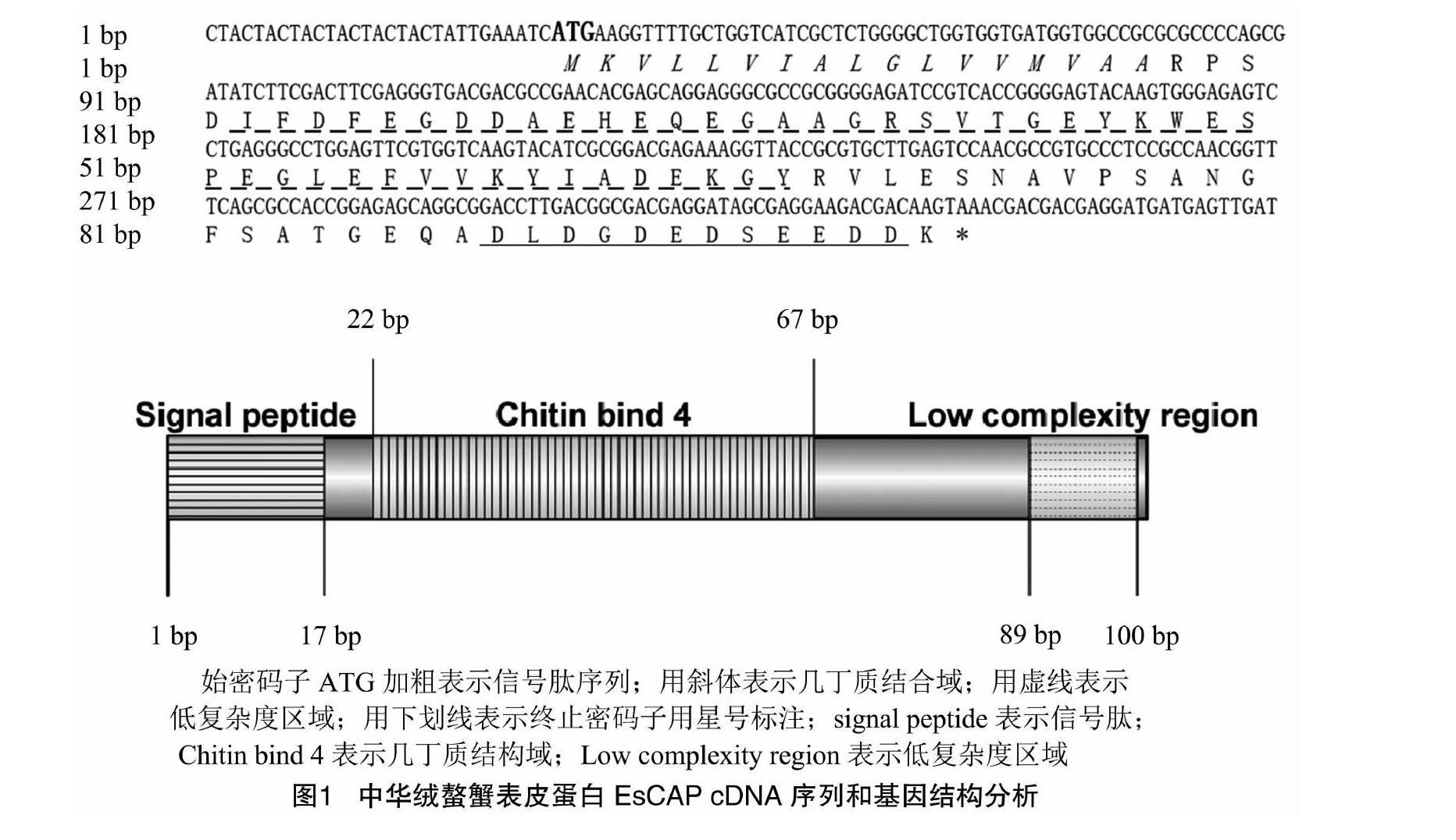
2.1 中华绒螯蟹表皮蛋白基因EsCAP cDNA的序列分析
序列结果显示,中华绒螯蟹表皮蛋白基因EsCAP cDNA序列全长380 bp,其中包括29 bp的5′端非编码区、45 bp 3′端非编码区和306 bp的开放阅读框,共编码101个氨基酸残基(GenBank登陆号为MT771647)。编码蛋白的理化性质分析预测该蛋白的相对分子质量为10.9 ku,理论等电点为387。蛋白质结构预测分析结果(图1)表明,中华绒螯蟹EsCAP氨基酸序列主要有1个信号肽、1个几丁质结合域ChtBD4和1个低复杂度区域。其中,几丁质结合域包含R&R保守序列Gx8Gx6YxAxExGYx7P(其中x表示多个氨基酸占据的位置,数字表示氨基酸的数目),表明EsCAP属于RR1亚族。此外,EsCAP氨基酸序列C端富含天冬氨酸、谷氨酸等酸性氨基酸,属于典型酸性蛋白。
2.2 中华绒螯蟹表皮蛋白EsCAP的同源性分析
由图2可知,与其他甲壳类表皮蛋白基因序列比较发现,中华绒螯蟹表皮蛋白EsCAP氨基酸序列与蓝蟹(Callinectes sapidus)、南美白对虾、克氏原螯虾(P. clarkii)、远海梭子蟹(Portunus pelagicus)的表皮蛋白相似度在40.6%~59.5%间。其中,中华绒螯蟹EsCAP与克氏原螯虾CAP2氨基酸序列同源性最高,为59.5%。采用Mega 6.0软件对包括中华绒螯蟹表皮蛋白EsCAP在内的12组表皮蛋白序列进行系统进化树的构建。使用软件中的Neighbor-Joining(NJ)方法,进行1 000次独立分析。系统进化树分析结果(图3)表明,中华绒螯蟹表皮蛋白EsCAP与其他虾蟹类酸性表皮蛋白聚类为一支,而昆虫类表皮蛋白聚为另一支,系统进化树表明EsCAP与虾蟹类CAP类表皮蛋白同源性较高。
2.3 中华绒螯蟹表皮蛋白EsCAP在不同组织中的表达
由图4可知,以中华绒螯蟹肌球蛋白β-actin为内参对照,通过RT-qPCR方法检测中华绒螯蟹表皮蛋白EsCAP的组织分布情况。结果表明,EsCAP在蜕皮间期不表达,主要在蜕皮后期的表皮组织中表达。
2.4 中华绒螯蟹表皮蛋白EsCAP在不同蜕皮时期的表达
选取中华绒螯蟹蜕皮前期(D)、蜕皮后期(A、B)、蜕皮间期(C)的表皮组织,以中华绒螯蟹肌球蛋白基因β-action为内参对照,通过RT-qPCR方法检测中华绒螯蟹表皮蛋白EsCAP基因在不同蜕皮时期的表达。由图5可知, 中华绒螯蟹表皮蛋白EsCAP进入到蜕皮前期(D)表达量较低,到蜕皮后期(A、B)高表达,在蜕皮间期(C)不表达。中华绒螯蟹表皮蛋白EsCAP在蜕皮时期的表达趋势与表皮形成及钙化时间相一致。
2.5 中华绒螯蟹表皮蛋白EsCAP在不同发育时期的表达
选取中华绒螯蟹受精卵、溞1期(ZⅠ)、溞3期(ZⅢ)、溞5期(ZⅤ)、大眼幼体和仔蟹1期共6个不同发育时期个体,以中华绒螯蟹肌球蛋白β-actin作为内参对照,通过RT-qPCR方法来检测EsCAP基因的表达趋势。由图6可知,表皮蛋白EsCAP基因在受精卵时期表达最低,进入到溞状幼体时期与大眼幼体时期表达量有所升高,到仔蟹1期表达量明显升高,EsCAP的表达量随着中华绒螯蟹发育阶段的推进而产生变化,总体呈升高的趋势。
3 讨论与结论
表皮蛋白与几丁质结合构成支撑机体抵御外界不良环境的外骨骼,在甲壳类的生长发育及蜕皮硬化中具有重要的作用[3]。甲壳类表皮蛋白数量庞大,根据其保守序列的不同,可分为CPR、CPAP、Crust-18、postmolt-18等家族,其中具有R&R保守结构域序列的CPR家族分布最为广泛,在甲壳类与昆虫中均存在[9-10]。本研究克隆获得的中华绒螯蟹表皮蛋白EsCAP含有与几丁质结合的R&R结构域,属于CRP家族中RR-1亚类,对中华绒螯蟹表皮的形成是至关重要的。EsCAP蛋白的N端与C端富含亲水氨基酸,如丙氨酸、缬氨酸、脯氨酸和甘氨酸,亲水性强,主要分布于未钙化的表皮,通过R&R结构域与几丁质相结合,多肽N端和C端区域延伸至纤维间隙,形成水填充的非共价键网络,对保持表皮的柔韧性不可或缺[5]。此外EsCAP蛋白的C端富含天冬氨酸、谷氨酸等酸性氨基酸,等电点为3.87,是个典型的酸性分子。酸性大分子在表皮的硬化过程中起着至关重要的作用[11-12]。在日本对虾(Penaeus japonicus)蜕皮后期尾扇表皮组织中克隆获得DD4(后来命名为Crustocalcin)和DD5酸性表皮蛋白,具有钙结合能力[13-14]。从克氏原螯虾表皮基质中分离纯化出2种钙化相关肽CAP-1和CAP-2,富含酸性氨基酸[15-16]。其中,CAP-1的钙结合能力明显大于CAP-2,这与CAP-1酸性氨基酸的数量多,在C末端分布更为集中有关[15-16]。中华绒螯蟹EsCAP氨基酸残基结构和分布与其他钙化相关肽相似,是否具有钙离子结合能力还有待于进一步试验验证。
虽然R&R保守结构域具有一定的保守性,但甲壳类表皮蛋白CAP在不同物种之间的相似度并不高。EsCAP与其他虾蟹类相似度在50%左右,与昆虫类相似度在30%左右(图2),这远低于同一亲缘关系水平的相似度。而近源的一些虾蟹类表皮蛋白CAP之间的相似度也不高,克氏原螯虾CAP-1与CAP-2的氨基酸相似度为46.2%。虽然表皮蛋白EsCAP的氨基酸残基相似性不高,但是该基因结构域保守序列位点非常保守。根据二级结构的预测,这些保守位点是R&R结构域形成β折叠构象的关键位点,影响着与几丁质结合[17-18]。
表皮蛋白基因的表達具有明显的组织分布差异性与时期特异性。中华绒螯蟹表皮蛋白EsCAP仅在蜕皮后期的表皮组织中高表达,而在其他时期以及其他组织中几乎不表达,具有明显的时空表达特异性(图4、图5)。这与蓝蟹表皮蛋白CAP、克氏原螯虾表皮蛋白CAP-2和CAP-1、日本对虾表皮蛋白DD4和DD5基因的表达趋势一致,均集中于蜕皮后期的表皮组织中高表达[11,13-16]。刚完成蜕皮后的甲壳类新表皮吸水后迅速延展,这一时期的表皮组织正伴随着表皮的快速扩张和钙化,具有几丁质结构域的表皮蛋白EsCAP可能通过R&R结构域与几丁质结合形成几丁质纤维复合物框架,参与新表皮的形成[19-20];与此同时,EsCAP中的酸性氨基酸与钙离子相结合,促进表皮组织碳酸钙沉淀,加速表皮钙化进程[21-22]。在不同发育时期,中华绒螯蟹表皮蛋白EsCAP在仔蟹1期的表达量最高,这与一些昆虫类RR1型表皮蛋白的表达趋势相似[23]。中华绒螯蟹从受精卵的孵化到仔蟹的发育,表皮的积累也越来越多,至仔蟹期表皮结构基本成型,表皮代谢需求量大,暗示表皮蛋白CAP可能参与不同发育阶段新表皮的合成。
中华绒螯蟹表皮蛋白EsCAP具有典型的R&R几丁质结合结构域,富含大量酸性氨基酸残基,主要在蜕皮后期的表皮组织中表达,推测EsCAP可能参与表皮的形成与钙化,其具体功能还有待于进一步验证。
参考文献:
[1]Delon I,Payre F. Evolution of larval morphology in flies:get in shape with shavenbaby[J]. Trends in Genetics,2004,20(7):305-313.
[2]Frédéric M,Luquet G. Unusually acidic proteins in biomineralization[M].Handbook of Biomineralization:Biological Aspects and Structure Formation,2008.
[3]Nagasawa H. The crustacean cuticle:structure,composition and mineralization[J]. Frontiers in Bioscience,2012,4(1):711-720.
[4]Roer R,Abehsera S,Sagi A. Exoskeletons across the pancrustacea:comparative morphology,physiology,biochemistry and genetics[J]. Integrative and Comparative Biology,2015,55(5):771-791.
[5]Andersen S O. Exoskeletal proteins from the crab,cancer pagurus[J]. Comparative Biochemistry and Physiology,1999,123(2):203-211.
[6]鄭征帆,吕艳杰,宁黔冀. 甲壳动物表皮几丁质结合蛋白结构与功能研究进展[J]. 水产科学,2017,36(4):538-542.
[7]Andersen O S. Biochemistry of insect cuticle[J]. Annual Review of Entomology,1979,24(1):29-59.
[8]2019年中国渔业统计年鉴[M]. 北京:中国农业出版社,2019.
[9]Andersen S O,Hjrup P,Roepstorff P. Insect cuticular proteins[J]. Insect Biochemistry and Molecular Biology,1995,25(2):153-176.
[10]Karouzou M V,Spyropoulos Y,Iconomidou V A,et al. Drosophila cuticular proteins with the R&R consensus:annotation and classification with a new tool for discriminating RR-1 and RR-2 sequences[J]. Insect Biochemistry and Molecular Biology,2007,37(8):754-760.
[11]Coblentz F E,Shafer T H,Roer R D. Cuticular proteins from the blue crab alter in vitro calcium carbonate mineralization[J]. Comparative Biochemistry and Physiology,1998,121(3):349-360.
[12]Glazer L,Sagi A. On the involvement of proteins in the assembly of the crayfish gastrolith extracellular matrix[J]. Invertebrate Reproduction & Development,2012,56(1):57-65.
[13]Endo H,Persson P,Watanabe T. Molecular cloning of the crustacean DD4 cDNA encoding a Ca2+-binding protein[J]. Biochemical and Biophysical Research Communications,2000,276(1):286-291.
[14]Endo H,Takagi Y,Ozaki N,et al. A crustacean Ca2+-binding protein with a glutamate-rich sequence promotes CaCO3 crystallization[J]. The Biochemical Journal,2004,384(1):159-167.
[15]Inoue H,Ohira T,Ozaki N,et al. Cloning and expression of a cDNA encoding a matrix peptide associated with calcification in the exoskeleton of the crayfish[J]. Comparative Biochemistry and Physiology(Part B Biochemistry & Molecular Biology),2003,136(4):755-765.
[16]Inoue H,Ohira T,Ozaki N,et al. A novel calcium-binding peptide from the cuticle of the crayfish,Procambarus clarkii[J]. Biochemical and Biophysical Research Communications,2004,318(3):649-654.
[17]Rebers J E,Riddiford L M. Structure and expression of a Manduca sexta larval cuticle gene homologous to drosophila cuticle genes[J]. Journal of Molecular Biology,1988,203(2):411-423.
[18]Rebers J E,Willis J H. A conserved domain in arthropod cuticular
proteins binds chitin[J]. Insect Biochemistry and Molecular Biology,2001,31(11):1083-1093.
[19]Noh M Y,Muthukrishnan S,Kramer K J,et al. Tribolium castaneum RR-1 cuticular protein TcCPR4 is required for formation of pore canals in rigid cuticle[J]. PLoS Genetics,2015,11(2):e1004963.
[20]Mun S,Noh M Y,Dittmer N T,et al. Cuticular protein with a low complexity sequence becomes cross-linked during insect cuticle sclerotization and is required for the adult molt[J]. Scientific Reports,2015,5:10484.
[21]Abehsera S,Weil S,Manor R,et al. The search for proteins involved in the formation of crustacean cuticularstructures[J]. Hydrobiologia,2018,825(1):29-45.
[22]Luquet G. Biomineralizations:insights and prospects from crustaceans[J]. ZooKeys,2012,176(176):103-121.
[23]Volovych O,Lin Z,Du J,et al. Identification and temporal expression profiles of cuticular proteins in the endoparasitoid wasp,Microplitis mediator[J]. Insect Science,2020,27(5):998-1018.
猜你喜欢 基因克隆 芒果MiCOL6基因的克隆及其生物信息学和表达分析热带作物学报(2020年4期)2020-08-06龙眼Myb-related-R1的克隆及生物信息学分析安徽农学通报(2020年9期)2020-08-03夏枯草PvGGPPS基因的克隆和诱导表达分析广西植物(2020年6期)2020-07-30甘草MYB转录因子GlMYB10的克隆与表达分析江苏农业科学(2019年17期)2019-11-13荸荠多酚氧化酶基因的克隆及其在鲜切荸荠贮藏过程中的表达分析江苏农业科学(2019年17期)2019-11-13紫参薯查尔酮合成酶及异构酶基因的克隆与表达分析热带作物学报(2018年5期)2018-05-14猪苓菌核2种热激蛋白基因的克隆及其表达分析中国中药杂志(2016年24期)2017-04-18泸宁鸡MyoD基因的克隆及生物信息学分析江苏农业科学(2016年9期)2016-11-28芒果UFGT基因的克隆及表达分析江苏农业科学(2016年5期)2016-07-23烟草电压依赖阴离子通道NtVDAC1.1的电子克隆及生物信息学分析山东农业科学(2015年11期)2016-01-11




