李卫英 辛静 赵文植 董章宏 马路遥 夏茂甜 高洁 辛培尧


摘 要:朱蕉(Cordyline fruticosa)是一種常见的园艺植物,广泛分布于亚洲、大洋洲、南美洲等热带地区。为了明确朱蕉叶绿体基因组特征,了解其系统发育地位,利用Illumina测序技术对其叶绿体基因组测序后,进行中线性绘图、ENC-plot、PR2-plot和SSR位点等相关数据的挖掘和分析。结果表明:朱蕉叶绿体基因组全长154 488 bp,GC含量为38%,其中大单拷贝区长85 279 bp,GC含量为36.1%,小单拷贝区长20 325 bp,GC含量为32.6%;双向重复区分别长24 442 bp,GC含量分别为43.4%。注释得到131种基因,其中unigenes的有111种,包括了78种蛋白编码基因,rRNA基因4种和tRNA基因29种。以朱蕉与龙舌兰科中19种植物的叶绿体基因组构建系统发育树,结果显示朱蕉与蓝朱蕉(Cordyline indivisa)的亲缘关系最近。朱蕉叶绿体基因组GC3、GC1与GC2的碱基含量分别为29.47%、46.45%和39.14%,GC3的碱基含量低于GC1和GC2,说明密码子末尾偏好以A、U结尾;ENC的取值范围为35.48~59.44,表明朱蕉密码子偏好性较弱;ENC-plot分析可知ENC频数比值在-0.05~0.05之间的有20个,表明密码子偏好性主要受自然选择压力的影响;PR2-plot分析中,T>A,G>C,说明朱蕉叶绿体基因组受多种因素的影响;以RSCU值和ENC值作为参考,确定17个密码子为最优密码子。在检测到的91个SSR位点中五核苷酸最少(3个),单核苷酸最多(55个),SSR位点中以A、T(A/T、AT/AT和AAAT/ATTT)为重复单元的占76.92%。说明朱蕉叶绿体基因组SSR位点多以A、T重复单元为主。研究结果可为朱蕉乃至朱蕉属植物分类及系统进化提供理论依据。
关键词:朱蕉;叶绿体基因组;系统发育;密码子中图分类号:S687.9 文献标识码:A
Genomic Characteristics and Phylogenetic Analysis of Chloroplast ofCordyline fruticosa
LI Weiying XIN Jing ZHAO Wenzhi DONG Zhanghong MA Luyao XIA Maotian GAO Jie XIN Peiyao
1. Southwest Research Center for Engineering Technology of Landscape Architecture (National Forestry and Grassland Administration), Southwest Forestry University, Kunming, Yunnan 650224, China; 2. Key Laboratory of National Forestry and Grassland Administration on Biodiversity Conservation in Southwest China, Southwest Forestry University, Kunming, Yunnan 650224, China; 3. Center for Integrative Conservation, Xishuangbanna Tropical Botanical Garden, Chinese Academy of Sciences, Mengla, Yunnan 666303, China
Abstract:
Cordyline fruticosais an common horticultural plant, widely distributed in tropical Asia, Oceania and South America. In order to clarify the characteristics of the chloroplast genome ofC. fruticosa and understand its phylogenetic status, the chloroplast genome was sequenced by Illumina sequencing technology, and the relevant data of middle linear plot, ENC-plot, PR2-plot and SSR loci were mined and analyzed. The total length of the complete chloroplast genome was 154 488 bp, and the GC content was 38%. The length of large single copy area was 85 279 bp and the GC content was 36.1%. The small single copy area was 20 325 bp, and the GC content was 32.6%. And bidirectional repetition zones were 24 442 bp, respectively, and the GC content was 43.4%. 131 genes were annotated, including 111 unigenes genes, including 78 protein-coding genes, 4 rRNA genes and 29 tRNA genes. Phylogenetic tree was constructed using the chloroplast genomes of 19 plant species inC. fruticosaand Agavaceae family.C. fruticosawas most closely related toC. indivisa. The content of GC3in the chloroplast genome ofC. fruticosa was 29.47%, GC1and GC2 were 46.45%, 39.14%, respectively. The base content of GC3was lower than that of GC1and GC2, indicating that codon ends preferred to end with A and U. The ENC values ranged from 35.48 to 59.44, indicating that the codon bias was weak. ENC-plot analysis showed that there were 20 ENC frequency ratios between–0.05 to 0.05, indicating that codon preference was mainly affected by natural selection pressure. PR2-plot analysis revealed T>A, G>C, indicating that the chloroplast genome ofC. fruticosawas affected by many factors. Codon preference is mainly influenced by selection. Using RSCU and ENC values as references, 17 codons were determined as optimal codons in the chloroplast genome ofC. fruticosa. Among the 91 SSR loci detected, there were 3 pentanucleotides and 55 single nucleotides. 76.92% of the SSR loci took A, T (A / T, AT / AT and AAAT / ATTT) as repeat units. It showed that the A and T repeat units were the main SSR sites in the chloroplast genome ofC. fruticosa. The results of this study could provide a theoretical basis for the taxonomy and phyletic evolution ofC. fruticosaandCordyline.
Keywords:
Cordyline fruticosa; chloroplast genome; phylogeny; codon
DOI:
10.3969/j.issn.1000-2561.2022.04.001
朱蕉(Cordyline fruticosa)为龙舌兰科(Agavaceae)朱蕉属(Cordyline)植物。朱蕉喜高温多湿气候,主要分布于亚洲、大洋洲和南美洲的热带地区[1]。其株形优美,色彩华丽高雅,具有较高的观赏性,同时还可用于服饰、食品包装[2]等方面,因此在中国南部和台湾等地区广泛种植[3]。此外,朱蕉也是一种药用植物[4],叶片中含有次生代谢物甾体皂苷,可用于抗菌、抑制肿瘤细胞、治疗腹泻等[5-6]。目前,关于朱蕉的相关研究仅见于组织培养[7-9]、病虫害治理[4, 10-11]、扦插繁殖[2-3, 11]、盐胁迫[12-14]和RAPD、SSR分子标记[15-17]等方面。
植物叶绿体是一些高等植物和藻类所特有的细胞器[18-19],是进行光合作用的重要场所[20]。叶绿体基因组具有保守的四分体结构,由大单拷贝区(large single copy, LSC)、2个双向重复区(inverted repeat, IR)和小单拷贝区(small single copy, SSC)组成[21]。叶绿体基因组具有相对独立的遗传系统、进化速率快、分子量相对保守、序列获得容易等特点,被广泛地应用于分子进化、DNA条形码开发、物种鉴定、物种演化和系统发育分析等方面[22-23]。其中系统发育分析在物种进化研究、植物分类和亲缘关系鉴定等方面备受科研工作者的关注,如在花园君子兰(Clivia gardenii)[21]、香花枇杷(Eriobotrya fragrans)[24]、羽叶丁香(Syringa pinnatifolia)[25]、毛重楼(Paris mairei)[26]和杜梨(Pyrus betulifolia)[27]等植物中的应用。此外,在细胞质中,通过mRNA翻译成蛋白质时,遗传密码子担当着重要的角色。编码同一种氨基酸的不同密码子,在遗传学中称之为同义密码子,不同的蛋白质编码在同义密码子的使用上是有所不同的,但同一物种或亲缘关系相近的物种间却有着相似的使用偏好性[28-29]。密码子偏性在植物類群相关研究方面备受关注,其在遗传改良和系统发育等方面具有重要的意义。目前关于叶绿体基因组特征分析已应用于多种植物,如金刚等[30]以剑麻H.11648(Agave sisalana)栽培种进行叶绿体基因组相关研究,结果表明其密码子偏好性是受自然选择压力和突变压力的共同影响。在香花枇杷(E. fragrans)[31]、蝴蝶兰(Phalaenopsis aphrodite)[32]和蒜头果(Malania oleifera)[33]等叶绿体基因组分析中也表明密码子使用偏好性的因素是多种因素共同决定的。
目前,龙舌兰科植物的叶绿体全基因组已在香龙血树(Dracaena fragrans)[34]、柬埔寨龙血树(Dracaena cambodiana)[35]、丝兰(Yucca flaccida)[36]和剑麻(A. sisalana)[30]等多种植物中展开研究,但朱蕉在该方面的相关研究还未见报道。对朱蕉叶绿体基因组特征、系统发育等信息进行分析,可为朱蕉乃至朱蕉属植物叶绿体基因组的研究与应用提供理论基础。
1 材料与方法
1.1 材料
研究材料来自于中国科学院西双版纳热带植物园。采摘朱蕉新鲜嫩叶,裹上锡箔纸放入液氮中,备用。
1.2 方法
1.2.1 基因组的测序和注释 采用宋国立等[37]的方法对朱蕉叶片的DNA进行提取和检测,从而形成测序文库,质检合格后采用Illumina Novaseq平台进行测序。由北京诺禾致源科技股份有限公司完成相关建库测序。
以蓝朱蕉(C. indivisa)叶绿体基因组(登录号:KX822776)为参照,由Getorganelle软件组装成完整的CP(叶绿体)基因组[38],使用在线软件GeSeq(https://chlorobox.mpimp-golm.mpg.de/ geseq.html)对朱蕉叶绿体基因组进行注释,使用Geneious 8.1软件对注释结果手动校正[39]。其结果已提交至樟科植物叶绿体基因组数据库(https:
//lcgdb. wordpress. com/)(登录号:LAU10100)。最后,利用OGDRAW(https://chlorobox.mpimp- golm. mpg.de/OGDraw. html)软件在线进行朱蕉叶绿体基因组的物理图谱的绘制。
1.2.2 系统发育分析 为研究朱蕉在龙舌兰科中的系统发育关系,以龙舌兰科中7个属植物作为主群体,分别为龙舌兰属(Agave)、朱蕉属(Cor?dyline)、晚芦荟属(Hesperaloe)、龙血树属(Dracaena)、虎尾兰属(Sansevieria)、丝兰属(Yucca)和Hesperocallis属。共有19个种,分别是美洲龙舌兰(A. americana KX519714)、剑麻H.11648(A. hybrid cultivarH.11648 MG642741)、灰叶剑麻(A. fourcroydesMW540496)、狭叶龙舌兰(A. angustifolia MW540498)、剑麻(A. sisalana MW540497)、Polianthes sp.(KX931464)、蓝朱蕉(C. indivisa KX822776)、柬埔寨龙血树(D. cambodianaMH 293451)、D. coohinchinensis(MN200195)、龙血树(D. dracoMN990038)、香龙血树(D. fragransMW123093)、金边虎尾兰(S. trifasciata var. laurentii MT922036)、短叶丝兰(Y. brevifolia MW281818, MW 281832)、H. parviflora(NC032703)、H. campanulate(NC032702)、H. undulata(NC032704)、Y. brevifolia×Y. jaegeriana(MW281839)、Y. jaegeriana(MW281848)。在百合科(Liliaceae)百合属(Lilium)中选择野百合(L. brownii)(登录号:KX822776)作为外类群进行系统发育分析。首先利用MAFFT(https://mafft.cbrc.jp/alignment/server/)软件进行在线序列比对,再使用BioEdit软件对矩阵进行手动校正,然后用CIPRES(https://www.phylo.org/ portal2/login!input.action)在线软件构建具有1000个Bootstrap的最大似然法(maximum likelihood, ML)系统发育树。最后使用FigTree软件对结果进行美化。
1.2.3 密码子相关参数计算 运用软件CodonW 1.4.2分析大于300 bp的47条CDS(蛋白质编码区),分析朱蕉叶绿体基因组各CDS的同义密码子相对使用度(relative synonymous codon usage, RSCU)和有效密码子数(Effective number of codon, ENC)。使用EMBOSS explorer中的CUSP(https://www.bioinformatics.nl/emboss-explorer/)在线软件分析获得朱蕉叶绿体基因组不同基因中GC1、GC2、GC3和GCall的碱基含量,并利用SPSS 25.0软件对其进行相关性分析。
1.2.4 朱蕉密码子使用偏好性影响因素相关分析 参考WRIGHT[40]提出的方法来判断朱蕉叶绿体基因组密码子使用的偏好性。在中性绘图分析中以GC3為横坐标、GC12(GC1与GC2的平均值)为纵坐标作散点图,对二者进行相关性分析,图中1个散点代表1个基因,分析GC1、GC2和GC3相关性。ENC-plot是以ENC值为纵坐标,GC3值为横坐标进行二维散点图的绘制,标准曲线公式为:ENC=2+GC3+29/(GC32+(1–GC3)2)。PR2-plot分析(Parity rule 2)是以G3/(G3+C3)值为横坐标,以A3/(A3+T3)值为纵坐标进行PR2-plot绘图分析。通过以上相关分析来揭示朱蕉叶绿体基因组密码子的使用偏好性,由此区分基因组密码子使用偏好性的情况和检测碱基组成对密码子偏好性之间的关系[41]。
1.2.5 最优密码子分析 参考WRIGHT[40]提出的方法从两端各选取10%的基因(前后各5条),建立高低基因表达库。若同一密码子既符合高表达密码子,又符合高频密码子,则将该密码子确定为叶绿体基因组的最优密码子。
1.2.6 朱蕉叶绿体基因组特征分析 利用MISA(https://webblast.ipk-gatersleben.de/misa/)在线软件进行朱蕉叶绿体基因组SSR(简单重复序列)序列的相关分析,参数设置为四核苷酸、五核苷酸和六核苷酸至少重复3次,三核苷酸至少重复4次,二核苷酸至少重复5次,单核苷酸至少10次。设置2个SSR位点之间的距离为100 bp,若2个位点间的距离小于100 bp,则2个序列组成1个复合微卫星。
2 结果与分析
2.1 朱蕉叶绿体基因组结构与特征
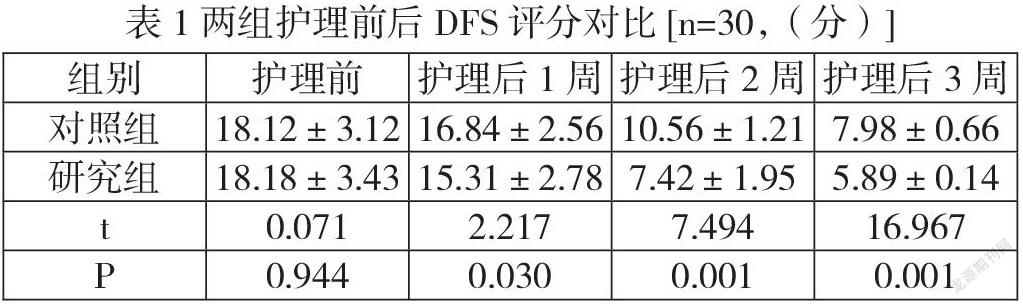
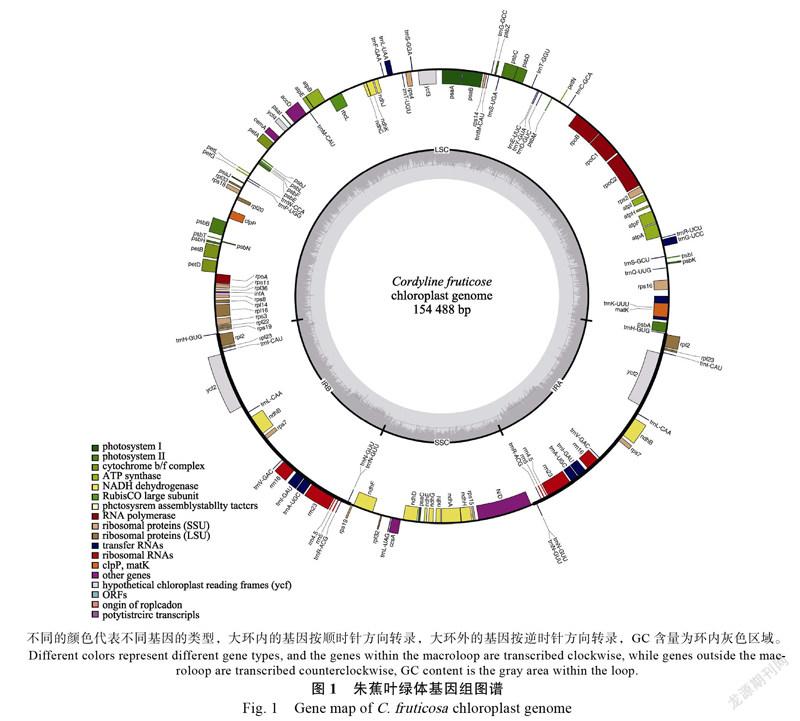
朱蕉叶绿体基因组全长为154 488 bp,GC含量为38.0%,是高度保守的典型四分体结构(图1),其中包括了大单拷贝区85 279 bp,GC含量为36.1%;小单拷贝区20 325 bp,GC含量为32.6%;双向重复区分别长24 442 bp,GC含量分别为43.4%。朱蕉叶绿体基因组共注释出131种基因,其中unigenes的有111种,分别为rRNA基因4种、编码蛋白质基因78种和tRNA基因29种(表1)。
2.2 系统发育分析
利用最大似然法(ML)对龙舌兰科中19种植物进行系统发育分析(图2),图中19种植物分为4个组:GroupⅠ(ML-100%)包括朱蕉属、龙血树属和虎尾兰属的7个种,分别是朱蕉(C. fruticosa)、蓝朱蕉(C. indivisa)、金边虎尾兰(S. trifasciata)、柬埔寨龙血树(D. cambodiana)、龙血树(D. draco)、香龙血树(D. fragrans)和D. coohinchinensis;GroupⅡ(ML-100%)包括晚芦荟属和Hesperocallis属共3个种,分别是H. parviflora、H. campanulate和H. undulata;GroupⅢ(ML-100%)包括丝兰属的3个种,分别是短叶丝兰(Y. brevifolia)、Y. brevifolia×Y. jaegeriana和Y. jaegeriana;GroupⅣ(ML-100%)包括龙舌兰属和晚香玉属中的6个种,分别是美洲龙舌兰(A. americana)、Polianthessp.、剑麻H.11648(A. hybrid cultivarH.11648)、灰叶剑麻(A. four-croydes)、狭叶龙舌兰(A. angustifolia)和剑麻(A. sisalana)。分析结果表明朱蕉与蓝朱蕉形成一个单系群,且亲缘关系最近,GroupⅠ、GroupⅡ、GroupⅢ和GroupⅣ 4个属植物以100%的支持率相互形成一个单系群,而这些单系群共同组成了龙舌兰科。
2.3密码子相关性分析
利用CUSP在线程序和CodonW 1.4.2软件对经过筛选过的朱蕉叶绿体基因组的47条蛋白质编码序列(CDS)进行分析,结果如表2所示:所有CDS的平均GCall含量为38.29%,GC1为46.45%,GC2为39.14%,GC3为29.47%,表明朱蕉叶绿体基因组密码子在不同位置的GC含量分布不均匀,其GC碱基含量存在差异。GC1、GC2的GC碱基含量要高于GC3的GC碱基含量,说明朱蕉叶绿体基因组密码子碱基偏好以A/U(T)结尾。在47条CDS朱蕉叶绿体基因组密码子中其ENC值介于35.48~59.44之间,平均值为48.4,结果表明朱蕉叶绿体基因组密码子的使用偏性为中等偏下。
由表3可知,GCall和GC1、GC2碱基含量呈极显著相关(P<0.01),與GC3碱基含量呈显著相关(P<0.05),GC1和GC2碱基含量呈极显著相关,GC1、GC2碱基含量和GC3碱基含量不显著相关,说明朱蕉叶绿体基因组第3位碱基与第1位、第2位碱基间有较大差异,第1位和第2位之间的相似度很高。ENC和GC3碱基含量呈极显著相关,与GC1、GC2碱基含量不显著相关。上述结果说明影响密码子使用偏好性的是第3位碱基的构成。但由于密码子数N和ENC的相关系数是0.140,并未达到显著水平,则表明该基因序列长度对密码子偏好性影响较弱。
朱蕉叶绿体基因组相关RSCU分析如表4所示,共有34个密码子的RSCU值>1,且以U结尾的密码子有16个,以A结尾的密码子有14个,以G结尾的密码子有3个,以C结尾的密码子有1个,A、U结尾较多。说明朱蕉叶绿体基因组以G、C结尾的密码子偏好较弱,多偏好于A、U结尾。
2.4 密码子使用偏好性影响因素
从图3可知,朱蕉叶绿体基因组密码子GC12的取值范围在0.427~1.288之间,GC3的取值范围在0.213~0.408之间,GC12和GC3的趋势线率(即回归系数)为0.2687,其相关系数R=0.0592,说明朱蕉密码子GC12和GC3的相关系数较低,GC3的碱基组成与GC1、GC2有差异,且GC的含碱基量在基因组中高度保守,结果表明自然选择压力对朱蕉叶绿体基因组密码子的影响较强。
ENC-plot图中,朱蕉中大部分基因分布在曲线下方(图4),说明朱蕉叶绿体基因组密码子的偏好性受自然选择压力的影响较大。从表5 ENC比值频数分布表可知:ENC比值分布在–0.05~ 0.05区间的有20个,与预期值ENC值较近,其余的基因则与预期ENC值较远,即离标准曲线较远,说明朱蕉叶绿体基因组密码子偏好性主要受自然选择压力的影响。
PR2-plot绘图分析(图5)的4个平面区域内的基因并未均匀分布,朱蕉叶绿体基因组中大多基因分布在下半部分,说明在碱基使用上T>A,G>C;即自然选择压力不是影响朱蕉叶绿体基因组的唯一因素,也会受自然选择压力之外的因素的影响。
2.5 最优密码子的确定
从表4可知朱蕉叶绿体基因组的高频密码子(RSCU>1)有34个,△RSCU≥0.08的高表达密码子有25个,将同时满足△RSCU≥0.08和RSCU>1的密码子确认为朱蕉叶绿体基因组最优密码子,共筛选出17个最优密码子(UCU、UGU、UUA、UAA、UUG、CCU、CGU、CGA、AUU、ACU、AGU、AAA、GUU、GCU、GGU、GUA、GAA)。其中有6个以A结尾,10个以U结尾,1个以G结尾。
2.6朱蕉叶绿体基因组SSR分析
使用MISA分析朱蕉叶绿体基因组的SSR碱基组成,共检测到91个SSR位点,其中五核苷酸最少仅有3个,四核苷酸有9个,三核苷酸有11个,二核苷酸有13个,而单核苷酸最多有55个。在所检测到的朱蕉叶绿体基因组SSR中以A/T、AT/AT和AAAT/ATTT为重复单元的占76.92%,说明朱蕉叶绿体基因组SSR位点多以A、T重复单元为主。
3 讨论
系统发育树显示,龙舌兰属与丝兰属、晚芦荟属和Hesperocallis属互为姐妹类群,且各属间、种间分支明确,这与KATE等[42]、MCKAIN等[36]的研究结果一致,本研究構建的系统发育树各分支节点以较高的支持率阐明了龙舌兰科各属间、种间的亲缘关系。ZHU等[35]通过叶绿体基因组序列建立了柬埔寨龙血树ML系统发育树,树图显示蓝朱蕉与柬埔寨龙血树同为龙舌兰科。黄威廉[43]、VERLOOVE等[44]在龙舌兰科族的分类及地理分布的相关研究中,证明朱蕉属与龙血树属、虎尾兰属同属于龙血树族(Draccaeneae),这些结果与本研究结果一致,有力地证明了本研究结果的可靠性。龙舌兰科系统发育ML树的4组分支都能以极高的支持率聚集且互为姐妹类群,证明了植物叶绿体基因组系统发育树的构建在物种鉴定方面的准确性。
同义密码子在蛋白质与基因组的联系中发挥着重要的作用[45],密码子偏好性受多种因素影响,自然选择压力和突变压力是主要的影响因素[46]。基因的使用频率会受突变压力或自然选择压力的影响,通常GC含量的变化被认为是基因突变的趋势[30]。通常氨基酸不受密码子第3位碱基的改变而改变,而且突变时所受的自然选择压力也较弱,因此,可将GC3当作分子密码子使用模式的重要依据之一[47]。通过对朱蕉叶绿体基因组密码子偏好性的相关分析发现,GC1与GC2呈极显著相关,GC3与GC1、GC2不具有相关性,表明GC含量在密码子的各个位置上呈不均匀分布,RSCU的结果分析也证明了这一观点。在对米槠(Castan?opsis carlesii)[48]、羽叶丁香(S. pinnatifolia)[25]、阳春砂(Amomum villosum)[49]、矮扁桃(Prunus tenella)[50]、柿(Diospyros kaki)[51]、永椿香槐(Cladrastis yunchunii)[52]、灯盏花(Erigeron breviscapus)[53]和杜梨(P. betulifolia)[19]等植物的相关研究中,也得到了一致的观点。
通过中线性、PR2-plot和ENC-plot绘图分析发现,朱蕉叶绿体基因组密码子偏好性主要受自然选择压力的影响,这在紫花苜蓿(Medicageo sativa)[54]、秋茄(Kandelia obovata)[55]、杜梨(P. betulifolia)[19]、降香黄檀(Dalbergia odorifera)[56]和永椿香槐(C. yunchunii)[52]等植物的相关研究中也得出一致的结论。而在阔叶猕猴桃(Actinidia latifolia)[57]、蝴蝶兰(P. aphrodite)[32]、油茶(Camellia oleifera)[58]、甜荞(Fagopyrum esculentum)[59]、香花枇杷(E. fragrans)[31]和圆锥南芥(Arabis paniculate)[60]等植物的研究中则发现,这些植物的叶绿体基因组密码子偏好性不仅受到自然选择压力的影响,而且还会受到突变压力的影响。可见,在不同的植物物种之间其叶绿体基因组密码子偏好性的主要使用模式之间存在着差异性,并且这些影响因素的作用也不尽相同。本研究筛选出朱蕉叶绿体基因组17个最优密码子,多数以A、U结尾,这与剑麻(A. sisalana)[30]、乳油木(Vitellaria paradoxa)[61]、杧果(Mangifera indica)[62]和香花枇杷(E. fragrans)[31]等植物的相关研究中最优密码子多以A、U结尾的结果相同,这可能是由于叶绿体基因组富含A、U(T)碱基,且植物叶绿体基因组密码子的进化相对保守所造成。
通过MISA在线软件对朱蕉叶绿体基因组核苷酸进行位点标记,研究发现朱蕉叶绿体SSR基因组碱基偏好多以A/T碱基为主。这与云南火焰兰(Renanthera imschootiana)[63]、草果(Amomum tsaoko)[64]、狗枣猕猴桃(Actinidia kolomikta)[65]、沙枣(Elaeagnus angustifolia)[66]、裂叶榆(Ulmus laciniata)[67]、金花茶(Camellia petelotii)[68]和薏苡(Coix lacryma-jobi)[69]等研究结果一致。叶绿体基因组在进化过程中具有保守性和单亲遗传等特点,且叶绿体SSR技术在系统发育、物种分类和遗传多样性等方面有明显的优势[70]。朱蕉叶绿体SSR位点的获得对于后期进行龙舌兰科相关物种鉴定、遗传图谱的构建和系统发育等方面具有重要意义。
4 结论
朱蕉叶绿体基因组是典型的四分结构,全长154 488 bp,GC含量为38%,系统发育分析显示朱蕉与蓝朱蕉为姐妹种关系,对朱蕉密码子使用偏好性进行分析得出其更多受自然选择压力的影响,通过对朱蕉叶绿体基因组的密码子分析得出最优密码子有17个,其中16个是以A、U结尾的,朱蕉叶绿体基因组的SSR相关分析表明朱蕉叶绿体基因组SSR位点多以A、T重复单元为主。分析结果可为后续开展朱蕉属植物系统发育研究、物种鉴定、基因工程等的相关研究提供理论参考。
参考文献
- 中国科学院中国植物志编辑委员会. 中国植物志[M](第十四卷). 北京:
科学出版社, 1980:
273.Editorial Committee of Flora of China, Chinese Academy of Sciences. Flora of China (Vol.14)[M]. Beijing:
Science Press, 1980:
273. (in Chinese) 王金全. 细叶朱蕉扦插繁殖技术研究[J]. 福建林业科技, 2012, 39(1):
89-91.WANG J Q. Study on cutting propagation technology of Cordyline fruticosa ‘Bella[J]. Fujian Forestry Science and Technology, 2012, 39(1):
89-91. (in Chinese) 陈少萍. 朱蕉栽培管理[J]. 中国花卉园艺, 2015(6):
27-29.CHEN S P. Cordyline fruticosa cultivation management[J]. China Flowers & Horticulture, 2015(6):
27-29. (in Chinese) 童依婷, 晏冬华, 彭文煊, 黄静华, 黄洪滨, 易润华. 朱蕉叶枯病菌的鉴定及生物学特性[J]. 广东海洋大学学报, 2016, 36(4):
89-95.TONG Y T, YAN D H, PENG W X, HUANG J H, HUANG H B, YI R H. Identification and biological characteristics of Fusarium verticillioides (Saccardo) nirenberg causing leaf blight on Ti plant (Cordvline fruticosa Chevalier)[J]. Journal of Guangdong Ocean University, 2016, 36(4):
89-95. (in Chinese) ROMUALD T, FOUEDJOU, REMY B, TEPONNO, LUANA Q, MASSIMO B, DEZEMONA P, LUCA A. VITALI, DENNIS F L A. TAPONDJOU, LUCIANO B. Steroidal saponins from the leaves of Cordyline fruticosa (L.) A. Chev. and their cytotoxic and antimicrobial activity[J]. Phytochemistry Letters, 2014(7):
62-68. DASH S K,PADHY S. Review on ethnomedicines for diarrhoea diseases from orissa:
prevalence versus culture[J]. Journal of Human Ecology, 2006, 20(1):
59-64. 高建莉. 组织培养快速繁殖朱蕉[J]. 云南农业科技, 2000(4):
29.GAO J L. Rapid propagation of Cordyline by tissue culture[J]. Yunnan Agricultural Science and Technology, 2000(4):
29. (in Chinese) 潘学峰, 王 毅, 王安石. 密叶朱蕉组培快繁技术研究[J]. 热带林业, 2009, 37(1):
22-24.PAN X F, WANG Y, WANG A S. Study on tissue culture and rapid propagation technology of Dracaena deremensis cv. Compacta[J]. Tropical Forestry, 2009, 37(1):
22-24. (in Chinese) 王丽萍, 邓泽周, 吴勇谋, 田丽华, 谢 健, 谢晓辉, 张和顺, 梁番土. 皇帝蕉组织培养与快速繁殖技术研究[J]. 中国南方果树, 2014, 43(2):
71-73.WANG L P, DENG Z Z, WU Y M, TIAN L H, XIE J, XIE X H, ZHANG H S, LIANG F T. Study on tissue culture and rapid propagation technology of Musa paradisiaca[J]. South China Fruits, 2014, 43(2):
71-73. (in Chinese) 张 炜. 高温引发朱蕉褐斑病[J]. 花木盆景(花卉园艺), 2004(9):
24.ZHANG W. High temperature causes brown spot of Cordyline[J]. Flower Plant & Penjing, 2004(9):
24. (in Chinese) 陈 宣, 云 勇, 姜殿强, 张 浪, 李海文. 海南6种切叶花卉植物橡胶林下套种的适应性研究[J]. 西部林业科学, 2015, 44(1):
103-108.CHEN X, YUN Y, JIANG D Q, ZHANG L, LI H W. Adaptability of six leaf cutting flower plants interplanted in Hainan rubber plantation[J]. Journal of West China Forestry Science, 2015, 44(1):
103-108. (in Chinese) 蔡金桓, 都成林, 薛 立, 叶自慧, 佘汉基, 李子华. 盐胁迫对4种园林植物光合特性的影响[J]. 西南林业大学学报(自然科学), 2017, 37(2):
30-34.CAI J H, DOU C L, XUE L, YE Z H, SHE H J, LI Z H. Effects of NaCl stress on photosynthetic characteristic of garden seedlings of 4 species[J]. Journal of Southwest Forestry University (Natural Sciences) , 2017, 37(2):
30-34. (in Chinese) 郑欣颖, 李鹏飞, 薛 立, 麦凝珺, 蔡金桓, 李秋静. 3种园林植物的抗盐生理研究[J]. 中南林业科技大学学报, 2017, 37(9):
62-67.ZHENG X Y, LI P F, XUE L, MAI N J, CAI J H, LI Q J. Study on salt resistance physiology of three garden plants[J]. Journal of Central South University of Forestry & Technology, 2017, 37(9):
62-67. (in Chinese) 邹晓君, 列志旸, 薛 立. 盐分胁迫对7种园林植物生物量的影响[J]. 中南林业科技大学学报, 2018, 38(3):
97-101, 128.ZOU X J, LIE Z Y, XUE L. Effects of salt stress on biomass of seven garden plants[J]. Journal of Central South University of Forestry & Technology, 2018, 38(3):
97-101, 128. (in Chinese) 王 健, 宋希强, 张 鑫, 唐虹霞. 基于RAPD与形态数据的朱蕉品种亲缘关系分析[J]. 中国农学通报, 2009, 25(24):
322-325.WANG J, SONG X Q, ZHANG X, TANG H X. Analysis of genetic relationships on Ti plant (cordyline fruticosa) cultivars based on the RAPD markers and morphological traits[J]. Chinese Agricultural Science Bulletin, 2009, 25(24):
322-325. (in Chinese) 龙 兴, 秦献泉, 方 仁, 邓 彪, 安振宇, 黄伟雄, 尧金燕. 广西野生蕉种质资源调查与鉴定[J]. 西南农业学报, 2017, 30(6):
1284-1293. LONG X, QIN X Q, FANG R, DENG B, AN Z Y, HUANG W X, YAO J Y. Investigation and identification of wild Musa germplasm resources in Guangxi province[J]. Southwest China Journal of Agricultural Sciences, 2017, 30(6):
1284-1293. (in Chinese) 冯慧敏, 陈 友, 李 博, 邓长娟, 武耀廷. 中国芭蕉属野生种表型性状和SSR多样性分析[J]. 热带作物学报, 2011, 32(4):
708-714.FENG H M, CHEN Y, LI B, DENG C J, WU Y T. The diversity study of wild Musa species in China by phenotypic characteristics and SSR molecular markers[J]. Chinese Journal of Tropical Crops, 2011, 32(4):
708-714. (in Chinese) SHINOZAKI K, OHME M, TANAKA M, WAKASUGI T, SUGIURA M. The complete nucleotide sequence of the tobacco chloroplast genome:
its gene organization and expression[J]. Plant Molecular Biology Reporter, 1986, 5(9):
2043-2049. 辛雅萱, 董章宏, 瞿绍宏, 刘 成, 叶 鹏, 辛培尧. 杜梨叶绿体基因组密码子偏好性分析[J]. 河北农业大学学报, 2020, 43(6):
51-59.XIN Y X, DONG Z H, QU S H, LIU C, YE P, XIN P Y. Analysis on codon usage bias of chloroplast genome in Pyrus betulifolia Bge.[J]. Journal of Hebei Agricultural University, 2020, 43(6):
51-59. (in Chinese) KRZYSZTOF B, BURCH-SMITH T M. Chloroplast signaling within, between and beyond cells[J]. Frontiers in Plant Science, 2015, 6(781):
78. 吴海红, 李 丹, 冯秀丽, 岳 玲, 赵兴华. 花园君子兰叶绿体基因组序列[J]. 分子植物育种, 2021, 3(9):
1-8.WU H H, LI D, FENG X L, YUE L, ZHAO X H. The complete chloroplast genome sequence of Clivia gardenii[J]. Molecular Plant Breeding, 2021, 3(9):
1-8. (in Chinese) 尹为治, 方 正, 黄良鸿, 龙文兴, 李佳灵. 海南2种龙脑香科植物叶绿体基因组密码子偏好性分析[J]. 林业调查规划, 2020, 45(6):
19-26.YIN W Z, FANG Z, HUANG L H, LONG W X, LI J L. Codon usage bias in chloroplast genomes of two species of Dipterocarpaceae in Hainan[J]. Forest Inventory and Planning, 2020, 45(6):
19-26. (in Chinese) 段义忠, 张 凯. 沙冬青属植物叶绿体基因组对比和系统发育分析[J]. 西北植物学报, 2020, 40(8):
1323-1332.DUAN Y Z, ZHANG K. Comparative analysis and phylogenetic evolution of the complete chloroplast genome of Ammopiptanthus[J]. Acta Botanica Boreali-Occidentalia Sinica, 2020, 40(8):
1323-1332. (in Chinese) DONG Z H, QU S H, LIU C, YE P, XIN P Y. The complete chloroplast genome sequence of Eriobotrya fragrans[J]. Mitochondrial DNA Part B, 2019, 4(2): 3549-3550. 张靖雯. 濒危植物羽叶丁香叶绿体全基因组及系统发育研究[D]. 杨凌:
西北农林科技大学, 2019.ZHANG J W. The complete chloroplast genome and phylogenetic analysis of the endangered species Syringa pinnatifolia(Oleaceae[D]. Yangling:
Northwest University of Agriculture and Forestry Science and Technology, 2019. (in Chinese) 江 媛, 杨青淑, 王 婧, 杨成金, 黄林芳, 杨 燕, 段宝忠. 毛重楼叶绿体基因组序列特征及其系统发育分析[J]. 中草药, 2021, 52(13):
4014-4022.JIANG Y, YANG Q S, WANG J, YANG C J, HUANG L F, YANG Y, DUAN B Z. Complete chloroplast genome of Paris mairei:
characterization and phylogeny[J]. Chinese Traditional and Herbal Drugs, 2021, 52(13):
4014-4022. (in Chinese) DONG Z H, QU S H, LI X H, YE P, LIU C, XIN Y X, XIN P Y. The complete plastome sequence of a subtropical tree Pyrus betulaefolia (Rosaceae)[J]. Mitochondrial DNA Part B, 2020, 5(1):
826-827. SUEOKA N. Directional mutation pressure and neutral molecular evolution[J]. Proceedings of the National Academy of Sciences, 1988, 85(8):
2653-2657. 高家翔, 胖铁良, 唐 征, 张 兰, 何承忠, 杨 凯. 早园竹叶绿体atpA基因序列及系统进化分析[J]. 西南林学院学报, 2009, 29(2):
31-36.GAO J X, PANG T L, TANG Z, ZHANG L, HE C Z, YANG K. Sequence and phylogenetic analysis of atpA gene in chloroplast of early garden bamboo[J]. Journal of Southwest Forestry University (Natural Sciences), 2009, 29(2):
31-36. (in Chinese) 金 刚, 覃 旭, 龙凌云, 王丽萍, 覃剑峰, 危丹妮, 陈 涛, 蔡中全. 剑麻叶绿体基因组编码序列密码子的使用特征[J]. 福建农林大学学报(自然科学版), 2018, 47(6):
705-710.JIN G, QIN X, LONG L Y, WANG L P, QIN J F, WEI D N, CHEN T, CAI Z Q. Characteristics of codon usage in the chloroplast protein-coding genes of Agave hybrid No.11648[J]. Journal of Fujian Agriculture and Forestry University (Natural Science Edition) , 2018, 47(6):
705-710. (in Chinese) 屈亚亚, 辛 静, 冯发玉, 董章宏, 瞿绍宏, 王海洋, 李 斌, 辛培尧. 香花枇杷质体基因组序列密码子偏性分析[J]. 西北林学院学报, 2021, 36(4):
138-144, 158.QU Y Y, XIN J, FENG F Y, DONG Z H, QU S H, WANG H Y, LI B, XIN P Y. Codon usage bais in chloroplast genome of Eriobotrya fragrans Champ. ex Benth[J]. Journal of Northwest Forestry University, 2021, 36(4):
138-144, 158. (in Chinese) 续 晨, 贲爱玲, 蔡晓宁. 蝴蝶兰叶绿体基因组密码子使用的相关分析[J]. 分子植物育种, 2010, 8(5):
945-950.XU C, BEN A L, CAI X N. Analysis of synonymous codon usage in chloroplast genome of Phalaenopsis aphrodite subsp. formosana[J]. Molecular Plant Breeding, 2010, 8(5):
945-950. (in Chinese) 原曉龙, 刘 音, 康洪梅, 陈中华, 李云琴, 王 毅. 蒜头果叶绿体基因组密码子偏好性分析[J]. 西南林业大学学报(自然科学), 2021, 41(3):
15-22.YUAN X L, LIU Y, KANG H M, CHEN Z H, LI Y Q, WANG Y. Analysis of codon usage bias in chloroplast genome of Malania oleifera[J]. Journal of Southwest Forestry University (Natural Sciences), 2021, 41(3):
15-22. (in Chinese) JIANG H, KANG Y, CHEN X, YANG X L, YANG X Q. The complete plastid genome sequence of Dracaena fragrans (L.) Ker Gawl. (Asparagaceae)[J]. Mitochondrial DNA Part B, 2021, 6(2):
653-655. ZHU Z X, MU W X, WANG J H, ZHANG J R, ZHAO K K, FRIEDMAN C R, WANG H F. Complete plastome sequence of Dracaena cambodiana (Asparagaceae):
a species considered “Vulnerable” in Southeast Asia[J]. Mitochondrial DNA Part B, 2018, 3(2): 620-621. MCKAIN M R, MCNEAL J R, KELLAR P R, EGUIARTE L E, PIRES J C, LEEBENS M J. Timing of rapid diversification and convergent origins of active pollination within Agavoideae (Asparagaceae) [J]. American Journal of Botany, 2016, 103(10):
1717-1729. 宋国立, 崔荣霞, 王坤波, 郭立平, 黎绍惠, 王春英, 张香娣. 改良CTAB法快速提取棉花DNA[J]. 棉花学报, 1998, 33(5):
50-52.SONG G L, CUI R X, WANG K B, GUO L P, LI S H, WANG C Y, ZHANG X D. A rapid improved CTAB method for extraction of cotton genom ic DNA[J]. Cotton Science, 1998, 33(5):
50-52. (in Chinese) JIN J J, YU W B, YANG J B, SONG Y, DEPAMPHILIS C W, YI T S, LI D Z. GetOrganelle:
a fast and versatile toolkit for accurate de novo assembly of organelle genomes[J]. Genome biology, 2020, 21(1):
241. KEARSE M, MOIR R, WILSON A, STONES H S, CHEUNG M, STURROCK S, BUXTON S, COOPER A, MARKOWITZ S, DURAN C, THIERER T, ASHTON B, MEINTJES P, DRUMMOND A. Geneious basic:
an integrated and extendable desktop software platform for the rganization and analysis of sequence data[J]. Bioinformatics, 2012, 28(12):
1647-1649. WRIGHT F. The ‘effective number of codons used in a gene[J]. Genome biology, 1990, 87(1):
23-29. DURET L, MOUCHIROUD D. Expression pattern and, surprisingly, gene length shape codon usage in Caenorhabditis, Drosophila and Arabidopsis[J]. Proceedings of the National Academy of Sciences, 1999, 96(8):
4482-4487. KATE H, MARK F. A chloroplast phylogeny of Agavaceae subfamily Chlorogaloideae:
Implications for the tempo of evolution on serpentine soils[J]. Systematic Botany, 2013, 38(4):
674. 黄威廉. 世界龙舌兰科族属分类及地理分布[J]. 贵州科学, 2016, 34(1):
1-4.HUANG W L. Classification and geographical distribution of Agave families and genera in the world[J]. Guizhou Science, 2016, 34(1):
1-4. (in Chinese) VERLOOVE F, PASCUAL M S. Notes on genuine Agave vivipara (Agavaceae), a poorly known Caribbean species, recently introduced in the Canary Islands (Spain)[J]. British Cactus and Succulent Society, 2021, 2021(39):
259-264. 张家榕, 雷万钧. 18种苔藓植物rbcL基因的密码子偏性及聚类分析[J]. 山西农业大学学报(自然科学版), 2020, 40(6):
1-12.ZHANG J R, LEI W J. Codon bias and cluster analysis of rbcL genes in 18 bryophytes[J]. Journal of Shanxi Agricultural University(Natural Science Edition) , 2020, 40(6):
1-12. (in Chinese) ROMERO H, ZAVALA A, MUSTO H. Codon usage in Chlamydia trachomatis is the result of strand-specific mutational biases and a complex pattern of selective forces[J]. Nucleic Acids Research, 2000, 28(10):
2084?2090. 吴妙丽, 陈世品, 陈 辉. 竹亚科叶绿体基因组的密码子使用偏性分析[J]. 森林与环境学报, 2019, 39(1):
9-14.WU M L, CHEN S P, CHEN H. Codon usage bias analysis of chloroplast genome of Bambusaceae[J]. Journal of Forest and Environment, 2019, 39(1):
9-14. (in Chinese) 江淑珍, 连 辉, 熊远芳, 张 赛, 陈世品. 米槠叶绿体基因组密码子偏好性分析[J]. 分子植物育种, 2021, 26(7):
1-12.JIANG S Z, LIAN H, XIONG Y F, ZHANG S, CHEN S P. Analysis of codon bias in chloroplast genome of Castanopsis carlesii[J]. Molecular Plant Breeding, 2021, 26(7):
1-12. (in Chinese) 马孟莉, 孟衡玲, 张 薇, 雷 恩, 卢丙越. 阳春砂叶绿体全基因组解析及系统发育研究[J]. 西北植物学报, 2020, 40(6):
978-986.MA M L, MENG H L, ZHANG W, LEI E, LU B Y. Whole genome analysis and phylogeny of chloroplasts in Amomum villosum[J]. Acta Botanica Boreali-Occidentalia Sinica, 2020, 40(6):
978-986. (in Chinese) 杨 斌, 孟庆瑶, 张 凯, 段义忠. 孑遗濒危植物矮扁桃叶绿体全基因组特征分析及亲缘关系鉴定[J]. 植物研究, 2020, 40(5):
686-695.YANG B, MENG Q Y, ZHANG K, DUAN Y Z. Analysis of chloroplast genome characteristics and phylogenetic identification of endangered plant Amygdalus nana[J]. Bulletin of Botanical Research, 2020, 40(5):
686-695. (in Chinese) 傅建敏, 索玉静, 刘慧敏, 谭晓风. 柿属植物叶绿体蛋白质编码基因密码子用法[J]. 经济林研究, 2017, 35(2):
38-44.FU J M, SUO Y J, LIU H M, TAN X F. Analysis on codon usage in the chloroplast protein-coding genes of Diospyros spp[J]. Non-wood Forest Research, 2017, 35(2):
38-44. (in Chinese) 李江飞, 原晓龙, 李熙颜, 王 瑜, 蔡年辉, 陈 诗, 许玉兰. 永椿香槐叶绿体基因组密码子偏好性分析[J]. 分子植物育种, 2021, 10(7):1-13.LI J F, YUAN X L, LI X Y, WANG Y, CAI N H, CHEN S, XU Y L. Analysis on codon usage bias of chloroplast genome in Cladrastis yungchunii X. W. Li & G. S. Fan[J]. Molecular Plant Breeding, 2021, 10(7):
1-13. (in Chinese) 李显煌, 杨生超, 辛雅萱, 屈亚亚, 杨琳懿, 冯发玉, 范 伟, 辛培尧. 灯盏花叶绿体基因组密码子偏好性分析[J]. 云南农业大学学报(自然科学), 2021, 36(3):
384-392.LI X H, YANG S C, XIN Y X, QU Y Y, YANG L Y, FENG F Y, FAN W, XIN P Y. Analysis of the codon usage bias of chloroplast genome in Erigeron breviscapus (Vant.) Hand-Mazz[J]. Journal of Yunnan Agricultural University (Natural Science), 2021, 36(3):
384-392. (in Chinese) 喻 凤, 韩 明. 紫花苜蓿叶绿体基因组密码子偏好性分析[J]. 广西植物, 2021, 18(8):
1-13.YU F, HAN M. Analysis of codon usage bias in the chloroplast genome of alfalfa (Medicago sativa)[J]. Guihaia, 2021, 18(8):
1-13. (in Chinese) 赵 森, 邓力华, 陈 芬. 秋茄叶绿体基因组密码子使用偏好性分析[J]. 森林与环境学报, 2020, 40(5):
534-541.ZHAO S, DENG L H, CHEN F. Codon usage bias of chloroplast genome in Kandelia obovata[J]. Journal of Forest and Environment, 2020, 40(5):
534-541. (in Chinese) 原晓龙, 李云琴, 张劲峰, 王 毅. 降香黄檀叶绿体基因组密码子偏好性分析[J]. 广西植物, 2021, 41(4):
622-630.YUAN X L, LI Y Q, ZHANG J F, WANG Y. Analysis of codon usage bias in the chloroplast genome of Dalbergia odorifera[J]. Guihaia, 2021, 41(4):
622-630. (in Chinese) 王 宇, 周俊良, 唐冬梅, 仲伟敏, 马玉华, 张 敏. 阔叶猕猴桃叶绿体基因组特征及密码子偏好性分析[J]. 种子, 2020, 39(5):
13-19.WANG Y, ZHOU J L, TANG D M, ZHONG W M, MA Y H, ZHANG M. Analysis of chloroplast genome characteristics and codon preference in Broad-leaf Kiwifruit[J]. Seed, 2020, 39(5):
13-19. (in Chinese) 王鹏良, 杨利平, 吴红英, 农有良, 吴双成, 肖玉菲, 覃子海, 王华宇, 刘海龙.普通油茶叶绿体基因组密码子偏好性分析[J]. 广西植物, 2018, 38(2):
135-144.WANG P L, YANG L P, WU H Y, NONG Y L, WU S C, XIAO Y F, QIN Z H, WANG H Y, LIU H L. Condon preference of chloroplast genome in Camellia oleifera[J]. Guihaia, 2018, 38(2):
135-144. (in Chinese) 罗 洪, 胡莎莎, 吴 琦, 姚慧鹏. 甜荞叶绿体基因密码子偏爱性分析[J]. 基因组学与应用生物学, 2015, 34(11):
2457-2464.LUO H, HU S S, WU Q, YAO H P. Analysis of Buckwheat chloroplast gene codon bias[J]. Genomics and Applied Biology, 2015, 34(11):
2457-2464. (in Chinese) 罗 茜, 方 正, 余 婕, 翁庆北, 朱 斌. 圆锥南芥叶绿体基因组特征及密码子偏好性分析[J]. 分子植物育种, 2021, 26 (07):
1-27.LUO X, FANG Z, YU J, WENG Q B, ZHU B. Analysis of chloroplast genome characteristics and codon usage bias of Arabis paniculata franch[J]. Molecular Plant Breeding, 2021, 26 (7):
1-27. (in Chinese) 原晓龙, 李云琴, 张劲峰, 王 毅. 乳油木叶绿体基因组密码子偏好性分析[J]. 分子植物育种, 2020, 18(17):
5658-5664.YUAN X L, LI Y Q, ZHANG J F, WANG Y. Codon usage bias analysis of chloroplast genome in Vitellaria paradoxa[J]. Molecular Plant Breeding, 2020, 18(17):
5658-5664. (in Chinese) 辛雅萱, 黎若竹, 李 鑫, 陈丽琼, 唐军荣, 屈亚亚, 杨琳懿, 辛培尧, 李云芳. 杧果叶绿体基因组密码子使用偏好性分析[J]. 中南林业科技大学学报, 2021, 41(9):
148-156, 165.XIN Y X, LI R Z, LI X, CHEN L Q, TANG J R, QU Y Y, YANG L Y, XIN P Y, LI Y F. Analysis on codon usage bias of chloroplast genome in Mangifera indica[J]. Journal of Central South University of Forestry & Technology, 2021, 41(9):
148-156, 165. (in Chinese) 辛 静, 辛雅萱, 董章宏, 瞿绍宏, 姚国琼, 李 凤, 屈亚亚, 唐军荣. 云南火焰兰转录组SSR分布及其序列特征分析[J]. 南方农业学报, 2020, 51(7):
1634-1641.XIN J, XIN Y X, DONG Z H, QU S H, YAO G Q, LI F, QU Y Y, TANG J R. Distribution and sequence characteristics of SSR in transcriptome of Renanthera imschootiana Rolfe[J]. Journal of Southern Agriculture, 2020, 51(7):
1634-1641. (in Chinese) 马孟莉, 张 薇, 孟衡玲, 卢丙越. 草果叶绿体基因组特征及系统发育分析[J]. 中草药, 2021, 23(9):
1-9.MA M L, ZHANG W, MENG H L, LU B Y. Characterization and phylogenetic analysis of the complete chloroplast genome of Amomum tsao-ko[J]. Chinese Traditional and Herbal Drugs, 2021, 23(9):
1-9. (in Chinese) 吴东洋, 业 宁, 徐逸卿, 张 礼, 周小亮. 狗枣猕猴桃叶绿体基因组微卫星特征分析[J]. 北方园艺, 2018(9):
30-35.WU D Y, YE N, XU Y Q, ZHANG L, ZHOU X L. Microsatellite analysis of chloroplast genome of Actinidia kolomikta[J]. Northern Horticulture, 2018(9):
30-35. (in Chinese) 王 婧, 王天翼, 王罗云, 张建国, 曾艳飞. 沙枣叶绿体全基因组序列及其使用密码子偏性分析[J]. 西北植物学报, 2019, 39(9):
1559-1572.WANG J, WANG T Y, WANG L Y, ZHANG J G, ZENG Y F. Assembling and analysis of the whole chloroplast genome sequence of Elaeagnus angustifolia and its codon usage bias[J]. Acta Botanica Boreali-Occidentalia Sinica, 2019, 39(9):
1559-1572. (in Chinese) 杜久军, 左力辉, 刘易超, 于晓跃, 董 研, 王进茂, 杨敏生. 裂叶榆叶绿体基因组及CP-SSR位点分析[J]. 植物遗传资源学报, 2018, 19(6):
1187-1196.DU J J, ZUO L H, LIU Y C, YU X Y, DONG Y, WANG J M, YANG M S. Chloroplast genome and CP-SSR site analyses of Ulmus laciniata[J]. Journal of Plant Genetic Resources, 2018, 19(6):
1187-1196. (in chinese) 叶 鹏, 李显煌, 唐军荣, 李 斌, 张贵良, 刘 成, 雷 瀚, 辛培尧. 云南金花茶转录组SSR的分布及其序列特征[J]. 中南林业科技大学学报, 2019, 39(9):
86-91.YE P, LI X H, TANG J R, LI B, ZHANG G L, LIU C, LEI H, XIN P Y. Distribution and characteristics of SSR in transcriptome of Camellia fascicularis[J]. Journal of Central South University of Forestry & Technology, 2019, 39(9):
86-91. (in Chinese) 李祥栋, 潘 虹, 陆秀娟, 魏心元, 陆 平, 石 明, 秦礼康. 薏苡叶绿体基因组的SSR位点分析及种质亲缘关系鉴定[J]. 贵州农业科学, 2018, 46(8):
1-5.LI X D, PAN H, LU X J, WEI X Y, LU P, SHI M, QIN L K. SSR locus analysis of chloroplast genome and identification of genetic relationship in 14 semen adlay germplasm resources[J]. Guizhou Agricultural Sciences, 2018, 46(8):
1-5. (in Chinese) PROVAN J, POWELL W, HOLLINGSWORTH P M. Chloroplast microsatellites:
New tools for studies in plant ecology and evolution[J]. Trends in Ecology and Evolution, 2001, 16(3):
142-147.